
2020년 지금 우리는 코로나의 시대에 노출되어 있다. 가볍게 지나갈것 같았던 코로나는 약 7개월동안, 그리고 앞으로 언제까지도 지속될지 이제는 모르는 상황이다. R공부를 하면서 Tidyverse를 알게 되었고, Covid19에 대해 찾아 보다가 다음의 패키지를 알게 되었다. tidycovid19라는 패키지인데, 찾아 보니 많은 데이터를 넣어서 잘 만들어 좋은 패키지라 본다. joachim-gassen이라는 사람이 만든 패키지로 이분은 이 패키지를 다음과 같이 이름을 지었다.
Download, Tidy and Visualize Covid-19 Related Data
깃헙 링크를 보면 더 자세하게 알수 있으며 이번 블로그는 깃헙에 적인 내용을 정리해보는 시간을 가져보려 한다.
- Github: joachim-gassen/tidycovid19
- 블로그 링크(같은 내용이지만 더 잘 되어 있음): tidycovid19
다른 Covid-19 R packages는?
- {nCov2019}: 이 패키지는 중국 데이터에 중점울 두었지만 중국을 외에도 다른 나라 데이터를 포함하고 있다.
- {coronavirus}: 이 패키지는 대시보드와 함께 Johns Hopkins University CSSE dataset를 제공한다.
또한, 추가적으로 관련 자료 링크 두가지를 소개 해주는데, 이 두개만 해도 많은 자료를 담고 있다.
Stats and R: Top 100 R resources on Novel COVID-19 Coronavirus
github 링크: mine-cetinkaya-rundel/covid19-r
Data
현재 이 패키지는 다음의 함수를 통해 데이터를 제공한다. 참고로, 이 패키지는 매일 갱신과 Merge를 같이 해주기에 맘에 든다.
download_jhu_csse_covid19_data(): 이 데이터는 바이러스의 전지구적 확산을 평가하는 데 관심이 있는 연구자와 일반 청중을 위한 표준 자료로 발전했다. 데이터는 국가 및 하위 국가 수준에서 제공된다.download_ecdc_covid19_data(): 이 데이터는 매일 업데이트 되고, 일별 및 국가별로 Covid-19의 사례에 대한 최신 데이터가 포함되어 있다.download_owid_testing_data(): Our world in Data 라는 팀이 수집한 데이터로, 여러 국가 로부터 Covid-19테스트에 대한 데이터를 체계적으로 수집한 데이터가 모여있다.download_acaps_npi_data(): acaps에서 만든 데이터로 비의약적인 상황에 의해 바이러스에 어떠한 영향을 미치는지에 대한 데이터가 포함되어 있다.download_oxford_npi_data(): -download_apple_mtr_data(): 애플의 Mobility Trends. 애플맵 관련한 데이터라고 한다.download_google_cmr_data(): 구글의 Mobility Trends. 애플과 비슷하게 Covid-19관련해서 유저들의 이동 추세를 표현해준다.download_google_trends_data(): “coronavirus”라는 용어에 대한 검색 데이터.- 링크: Google Trends
download_wbank_data(): “World Bank”에서 제공 하는데이터. 특히 R에서 wbstats라는 패키지로도 사용이 가능하다.- 링크: World Bank
download_merged_data(): 위의 모든 데이터를 Merge한 데이터. 이 {tidycovid19} 패키지의 핵심 요소.
패키지 설치
remotes::install_github("joachim-gassen/tidycovid19") 코드를 사용해서 패키지 설치를 해주면 된다.
어떻게 사용할 수 있을까
tidycovid19 패키지 에서는 많은 함수를 제공해주는데, 다음 코드만 봐도 쉽게 시각화를 할 수가 있다.
library(tidyverse)
library(tidycovid19)
library(zoo)
df <- download_merged_data(cached = TRUE, silent = TRUE)
df %>%
filter(iso3c == "ITA") %>%
mutate(
new_cases = confirmed - lag(confirmed),
ave_new_cases = rollmean(new_cases, 7, na.pad=TRUE, align="right")
) %>%
filter(!is.na(new_cases), !is.na(ave_new_cases)) %>%
ggplot(aes(x = date)) +
geom_bar(aes(y = new_cases), stat = "identity", fill = "lightblue") +
geom_line(aes(y = ave_new_cases), color ="red") +
theme_minimal()
시각화
tidycovid19 패키지는 양질의 데이터를 수집하여 이를 한데 모아논것 뿐만 아니라 시각화를 해주는데도 뛰어나게 사용을 할 수가 있다. 다음 소개 해주는 몇가지의 함수와 옵션만 확인해도 세게의 Covid19에 대한 기본적인 파악이 가능하다.
시간에 따른 Corona에 의한 사망 시각화
다음은 plot_covid19_spread()함수를 사용해, 시간에 따른 corona에 의한 사망을 시각화 한것이다. 상당히 간단하며, 이 함수에 대해 좀 더 깊이 알고 싶으면 다음의 링크를 확인하자.
merged <- download_merged_data(cached = TRUE, silent = TRUE)
plot_covid19_spread(
merged, highlight = c("ITA", "ESP", "GBR", "FRA", "DEU", "USA", "KOR","CHN","JPN"),
intervention = "lockdown", edate_cutoff = 60
)
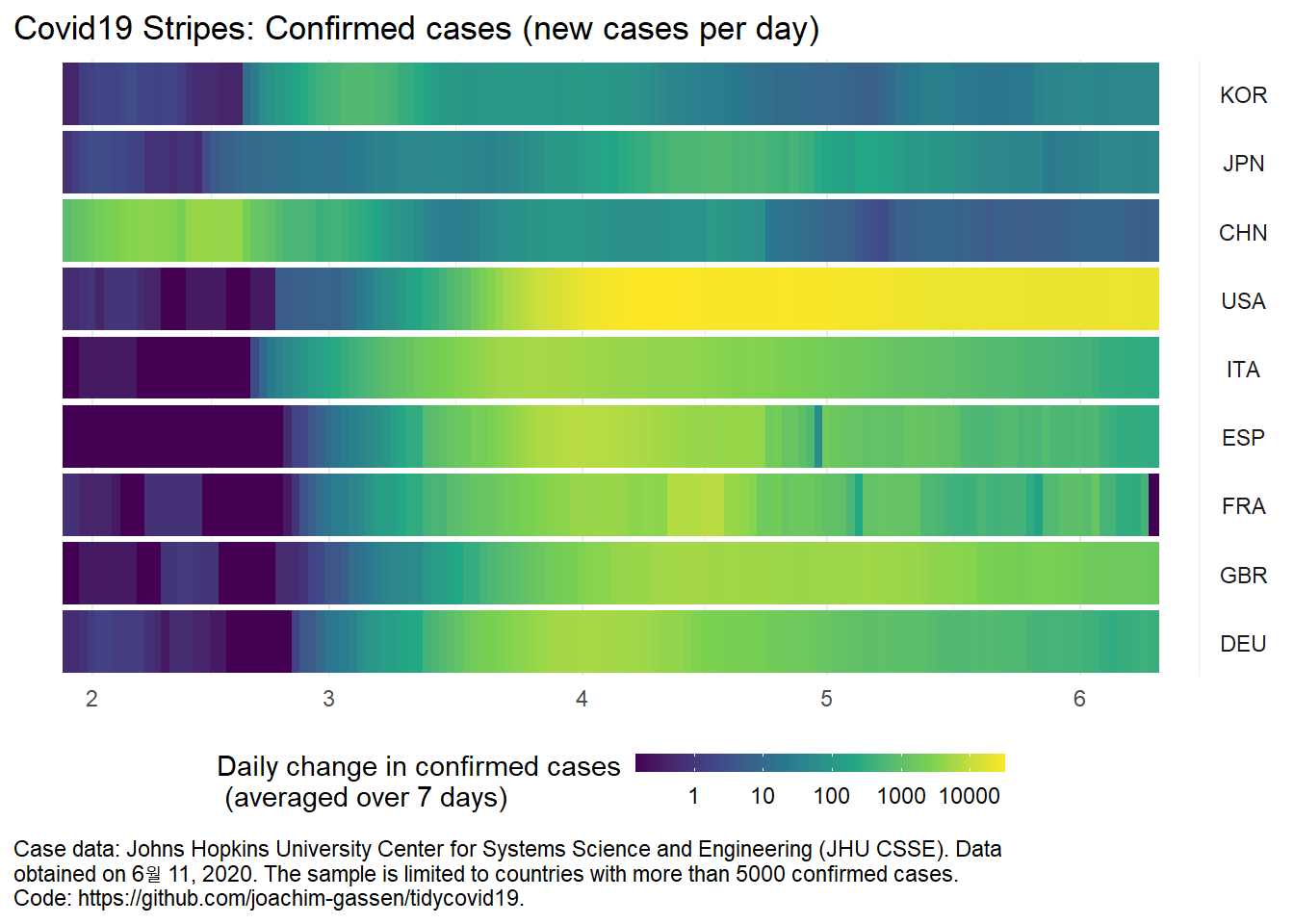
Covid 19 확산의 또다른 시각화
코로나의 확산을 시각화 하는 다른 방법. plot_covid19_stripes()함수를 사용하면 되고 자세한 옵션은 다음 링크에 잘 나와 있다.
plot_covid19_stripes(
type = "confirmed",
countries = c("KOR", "JPN", "CHN", "USA", "ITA", "ESP", "FRA", "GBR", "DEU"),
sort_countries = "countries"
)
Covid-19 지도
지도 데이터도 제공을 해준다. 함수 한방에. 기존에 지도를 뽑으려면 여러 조건들이 있고, 배워야 할게 많은데 이점은 정말 맘에 든다. map_covid19()함수를 사용하며 이 코드에 대해 자세히 알고 싶으면 다음 링크를 확인하자.
코로나 세계 지도 (누적 사망)
map_covid19(merged, cumulative = TRUE)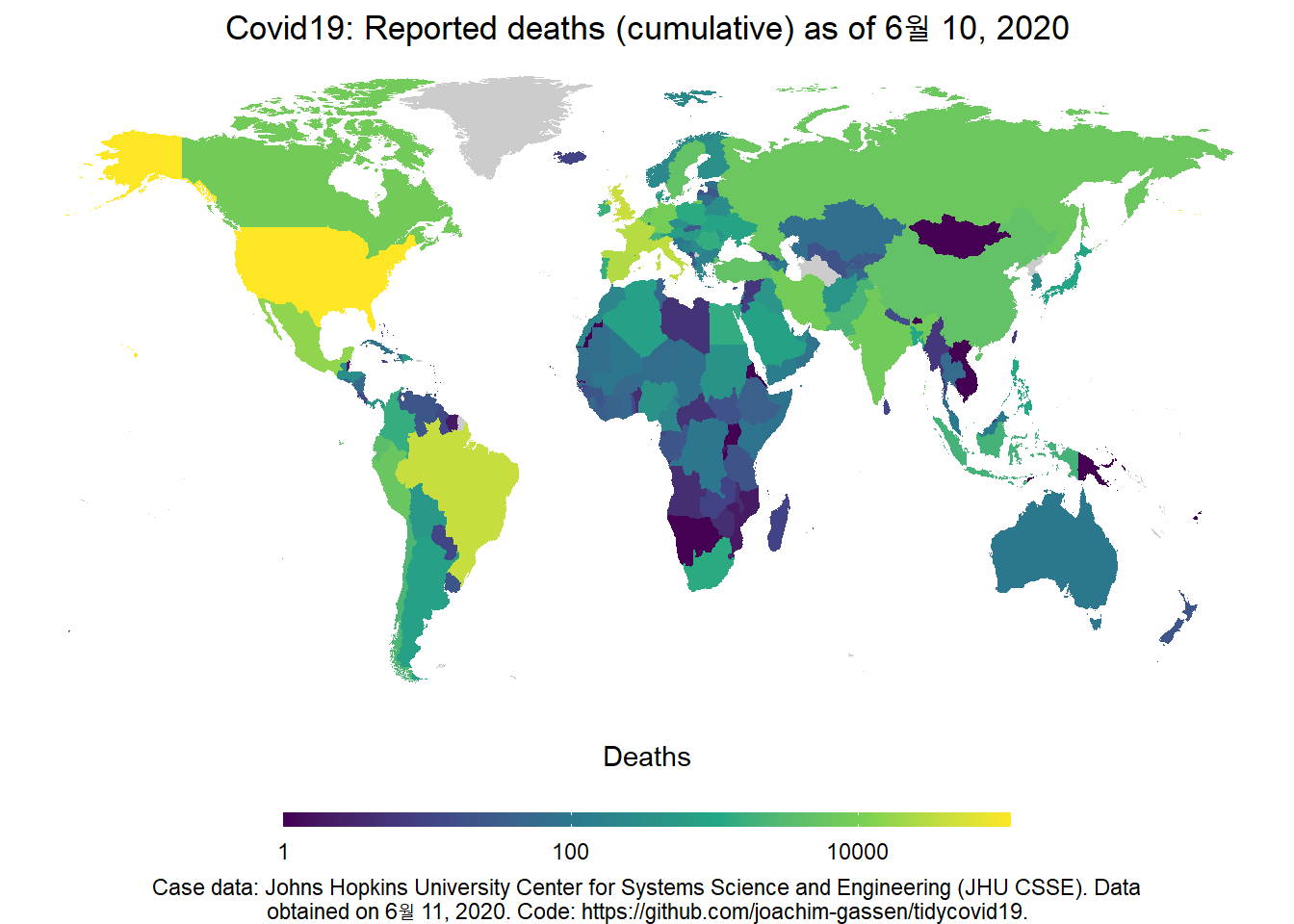
코로나 아시아 지도(누적 사망)
다음은 아시아만 따로 뽑아서 코로나 지도를 만든 모습.
map_covid19(merged, type = "confirmed", region = "Asia") 
코로나 애니메이션
정말 마음에 든다. 애니메이션 함수까지 제공을 해준다. Rendering 문제로 이곳에는 이렇게 코드만 남겨두도록 하자.
map_covid19(merged, type = "confirmed", dates = unique(merged$date))
Shiny App
샤이니 앱까지 제공을 해주는거 같다… shiny_covid19_spread() 인데 조금 더 파악이 필요하다. 원문은 다음과 같은데, shiny_covid19_spread()함수와, rclpboard 패키지로 쉽게 온라인에 앱을 띄울수가 있다고 한다.
Sorry, I could not resist. The options of the plot_covid19_spread() make the implementation of a shiny app a little bit to tempting to pass. The command shiny_covid19_spread() starts the app. Click on the image to be taken to the online app. You can use it to customize your plot_covid19_spread() display as it allows copying the plot generating code to the clipboard, thanks to the fine {rclipboard} package. You can now also customize the app by providing plot_covid19_spread() options as a list to the plot_options parameter.
총평
정말 마음에 든다. joachim-gassen라는분이 만들어서 어떤 분인가 깃허브를 찾아보고, 뭐하는 분이신지 찾아 봤는데 딱히 정보가 없었다. 이 패키지를 만드느라 했던 그 노력이 정말 배워야 하지 않을까 하는 생각을 많이 하게 된다. 단순하게 좋은말 밖에 나오지 않는, 정말 좋은 패키지. 지도며 애니메이션이며 블로그만 보고 쉽게 Report를 할 수 있도록 사용자 친화적이게 잘 만들었다.
Reference
tidycovid19를 찾아보면서 여러 블로그를 확인 하게 되었다. 거진 비슷하지만 그래도 이분의 깃헙이랑 블로그를 보면서 공부하는게 가장 좋아 보인다.